第50回 新たなゲノム編集手法につながる現象を解明
1.はじめに
本年6月、米国や日本の研究者らは、大腸菌中で特定の酵素が働くことによりDNAが組み換わる仕組みを解明したとして、Nature誌に発表した。この現象を用いることで、既存の手法より優れたゲノム編集手法の開発につながる可能性があると注目されている。今回はそれについて、経緯や周辺状況等も踏まえ紹介する。
2.今回の研究を巡る経緯
(1)ゲノム編集方法の開発経緯
今回の研究成果は、新世代のゲノム編集の開発につながる可能性のあるものである。そこで、ゲノム編集を巡るトピックについてはこれまでも紹介してきたが、今一度簡単に復習しておく。
ゲノム編集技術は、ゲノムの中のターゲットとなる部分にピンポイントで作用し、そこでDNA切断することができる技術として開発されてきた。1996年にはZFN、2010年にはTALENという方法が開発された。
だが、現在圧倒的に使用されているのは2012年に開発されたCRISPR/Casという方法であり、特にCas9を用いた方法が汎用化している。これは各遺伝子に結び付くRNAと、切断を行うCasタンパク質を組み合わせたもので、もともと自然界にあった「細菌の獲得免疫機構」を利用したものだった。その功績により、2020年、同技術を開発した研究者らにノーベル化学賞が与えられた。
その改良として、たとえば、本ニューズレター(第10回 一塩基編集技術による遺伝子治療の幕開け)でも紹介したが、一塩基編集という、ゲノム中の一つの塩基を置換する技術も開発された。
遺伝子編集技術に残された課題としては、長いDNA配列を特定の場所に挿入したり切り出したりすることだった。CRISPR/Casはピンポイントでゲノムの小さなセグメント(部分)を書き換えるのに使用できるが、長い断片を自由に切り貼りできるまでにはなっていなかった。
(2)ゲノム編集による長いDNA配列の操作に対する開発状況
しかし、CRISPR/Casを改良することで、長いDNA配列の操作についても進展が見られてきている。
本ニューズレター(第40回 2024年に注目すべき4つのバイオテクノロジー)でも紹介したが、米国スタンフォード大学の研究者らは、DNA組換えを媒介する一本鎖アニーリングタンパク質(SSAP)をCRISPR/Casシステムと組み合わせ、ヒトゲノム中に最大2キロのDNAを導入することを示した。
また、プライム編集というゲノム編集技術にも注目が集まる。これは、酵素を選択的に呼び寄せる短いランディングパッドという配列を用いて大きな断片を染色体に挿入するものである。
米国マサチューセッツ工科大学の研究者らはこれを用いて、最大36キロベースのDNAを正確に導入できる部位特異的ターゲティングエレメント(PASTE)による導入を初めて行った。PASTEは患者から細胞を取り出して培養して修復するのに特に有望であり、臨床研究も行われつつある。
ただ、これらはまだ開発途上の技術であり、汎用化はできていない。通常の方法を用いるなら、組み込めるDNAはせいぜい100~200塩基対である。また、これらについては特に医療応用を考えた場合、他にも課題がある。
これらのCRISPR/Casを応用したゲノム編集では、最初にDNAを破壊し、次に細胞が元々有しているDNA修復システムにより、目的の変更が行われる必要がある。しかし、修復時に、意図しない遺伝子損傷が発生する可能性がある。研究レベルではまだしも、医療応用になるとそれは致命的になる。
また、ゲノムの大規模な操作を行うためにCRISPRベースのシステムを用いた取り組みでは複数のタンパク質、又はCas酵素と別のタンパク質との融合が必要になることが多い。そうなると、ゲノム編集に複数のステップが必要になり、複雑になる。
また遺伝子治療を行う場合、ウイルスやカプセルに必要な部品を組み込んで投与し、目的の部位に届ける必要があり、それでは容量が大きくなりすぎる可能性も懸念される。
これらの課題を全て克服できるような、全く新しいタイプのゲノム編集技術が求められているのである。
(3)「動く遺伝子」と大腸菌での編集現象
その突破口となりそうなもの、それは「動く遺伝子」である。
その発見はかなり昔~1940年代に遡る。米国のマクリントック(Barbara McClintock, 1902年~1992年)という女性科学者が、トウモロコシの葉っぱに斑(ふ)という白い部分がなぜできるのかを調べる過程で、その原因遺伝子が、その遺伝子がある染色体上で、あるいは他の染色体へと移動することを発見した。
この遺伝子は、「転移する」ということで「トランスポゾン」と命名された。彼女の発見は一見信じがたく、なかなか評価されなかったが、1983年になってやっと、「可動遺伝因子の発見」としてノーベル生理学・医学賞が授与された。
その後、そうした動く遺伝子は細菌等にも備わっていることが分かった。ただ、それがどのような仕組みで起こるかはこれまであまりよく分かっていなかった。また、そうした現象をゲノム編集と結びつけて考える研究者はほとんどいなかった。
3.今回の研究成果
今回の研究は、Nature誌及びNature Communication誌に、3報の論文として掲載された。
今回の一連の研究を行ったのは、米国アーク研究所や日本の研究者(西増弘志東京大学先端科学技術研究センター教授)らだった。彼らは、ゲノム編集への応用も視野に入れつつ、粘り強く、動く遺伝子の現象の解明に努めてきたのである。
「動く遺伝子」は、いずれも組み換えるための酵素遺伝子のセットが存在し、リコンビナーゼ、インテグラーゼ、トランスポザーゼと呼ばれている。これらの酵素は、互いに配列は異なるが、どれもDNA断片を組み換える機能をもっている。
研究者らは、ゲノム編集のツールを探すために、上記のさまざまな酵素を精査した。そして、そのうちIS110と呼ばれる転移因子ファミリーに的を絞り、電子顕微鏡等も用いて詳しく調べた。
その結果、IS110ファミリーの酵素は、標的に対して実に巧妙なシステムを用いていることが分かった。
組み換えに関わる部分から、組み換えを促す酵素リコンビナーゼと組み換える場所を特定するRNAが作られており、そのRNAの一方の端はゲノムに挿入される外来DNAの一部に結合し、もう一方の端はゲノム内の外来DNAが挿入される場所に結合することが分かった。そして同RNAはリコンビナーゼを標的部位に誘導し、あらかじめ決められた編集を促進するブリッジとして働くのである。このためこのRNAは「ブリッジRNA」と名付けられた。
大腸菌は、動く遺伝子による組換えをランダムに行っているのではなく、この酵素とRNAを働かせることで、必要な部分だけを狙った場所に正確に行っていることが明らかになった。
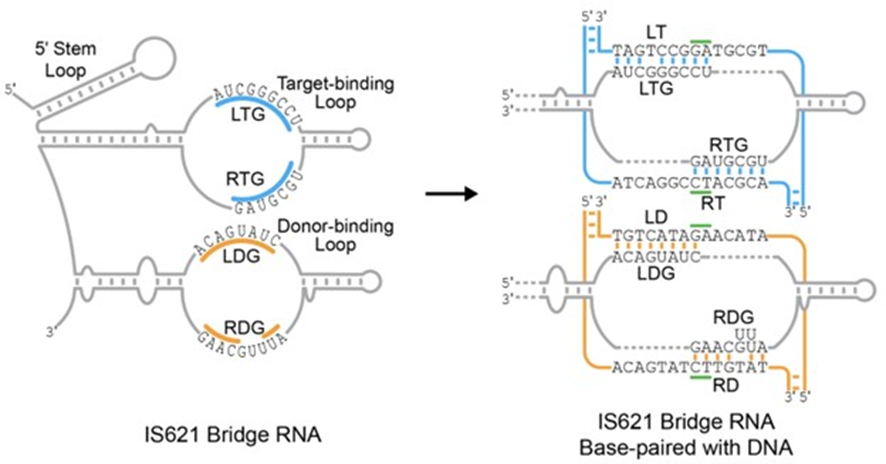
右図:同RNAのそれぞれのループがゲノム中の標的DNA及びドナーDNAと結びついている様子
(出典:Genetic Engineering and Biotechnology News)
そして研究者らは、このブリッジRNAの両端の配列を変更することで、IS110酵素を用途に沿って改変し、ゲノム内の希望する場所に任意の外来DNAを挿入することができた。彼らは、このシステムを用いて約5000塩基長のDNA片を大腸菌のゲノムに正確に挿入した。その際、大腸菌ゲノムから別のDNA断片を切り取って反転させた。
また別の研究では、IS110分子と同様のメカニズムを使用し、やはりプログラム可能なIS1111と呼ばれる別のファミリーの分子の生化学的特性を明らかにした。このRNA中間体はseekRNAと呼ばれる。
なお、西増弘志東京大学先端科学技術研究センター教授らは、低温電子顕微鏡を用いてリコンビナーゼの構造を探索し、その作用機序を詳細に調べることで、今回の現象解明に大いに貢献した。

同センターHPより引用
4.研究の意義と課題
今回の研究は、通常のCRISPR/Casシステムでの可能な範囲をはるかに超える、数千~数万塩基もの遺伝子改変を行える。またIS110及びIS1111のシステムでは、1つのタンパク質が必要なだけであり、これはCRISPR編集で使用される多くのCas酵素の半分以下のサイズである。またそのために実用化された場合には取り扱いが簡単で、さらにDNAを壊すことなくゲノムを操作できるため安全であり、医療応用には最適であろう。
ただし、問題点としてはこれがヒトに適用できるかどうかである。これまでのところ、IS110ファミリーの酵素-RNAは細菌や試験管内で実証されただけである。哺乳類細胞ではうまく機能しないという報告もある。だが、研究チームは現在、これらの酵素を哺乳類でうまく機能するように改変しようとしている。なお米国のバイオテクノロジー企業るサリオジェン・セラピューティクス社は、哺乳類トランスポゾンを用いたゲノム編集ツールの開発を目指している。
5.おわりに
ゲノム編集技術は、遺伝子を正確に操作する技術として知られているが、実際には完全に自由自在に操作できるという段階ではない。
しかし、世界の研究者による不断の研究の結果、改良を重ねられたり、今回のように全く新たな手法の発見・発明があったりすることにより、その進化はやまない。同技術を用いることで、生命を精密機械にたとえて一つ一つの部品の役割を解明し、生命の深い理解につながること、またそれが各分野に応用されていくことが期待される。
なお、ゲノム編集につながる細菌の獲得免疫現象を最初に発見したのは日本の研究者だとされているが、その後、ゲノム編集の進展にける日本の役割はそれほど大きなものとは言えなかった。かかる基盤技術の開発は広い分野の科学技術の進展につながるため、国際協力も必要である一方、知的所有権を通じた国益にもかかわる。
今回の現象の機構解明等において、日本の研究者が大きな役割を果たしているのは喜ばしいが、今後もそうした国際競争や国際協調の観点も含め、ゲノム編集技術の進展はしっかりフォローしていきたい。
参考文献
・H. Ledford “No CRISPR:‘ jumping gene’ enzyme edits genomes without breaking DNA”, (2024/6/27) Nature HP
(https://www-nature-com.translate.goog/articles/d41586-024-02141-x?error=cookies_not_supported&code=ebc3ce8f-35ac-4254-b76c-80f3b29e83f1&_x_tr_sl=en&_x_tr_tl=ja&_x_tr_hl=ja&_x_tr_pto=sct )
・J. D. Grinstein “Come together: Bridge RNAs close the gap to genome design”,(2024/06/26)Genetic Engineering and Biotechnology News
(https://www-genengnews-com.translate.goog/topics/genome-editing/come-together-bridge-rnas-close-the-gap-to-genome-design/?_x_tr_sl=en&_x_tr_tl=ja&_x_tr_hl=ja&_x_tr_pto=sc)
・M. L. Page “‘Bridge editing’ could be even better at altering DNA than CRISPR”, (2024/6/26) New Scientist(https://www.newscientist.com/article/2437237-bridge-editing-could-be-even-better-at-altering-dna-than-crispr/)
・東京大学先端科学技術研究センターHP 研究者紹介 014西増弘志 https://www.rcast.u-tokyo.ac.jp/ja/research/frontrunner_nishimasu.html
ライフサイエンス振興財団嘱託研究員 佐藤真輔

